Investigadores desvelan las claves genéticas que subyacen a la emergencia del cólera
30 junio 2025
Un nuevo estudio del Grupo de Genómica y Evolución Microbiana de la Universidad Miguel Hernández de Elche (UMH), junto con el Departamento de Interacciones hospedador-Microorganismo del Hospital St. Jude (Memphis, EE.UU.), arroja luz sobre uno de los grandes enigmas de la microbiología: por qué sólo algunas cepas de bacterias comunes se convierten en agentes pandémicos. 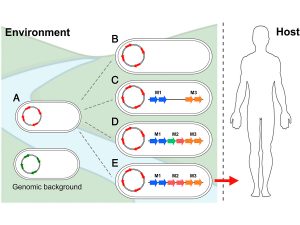 El trabajo, publicado en la prestigiosa revista científica PNAS, se centra en Vibrio cholerae, la bacteria causante del cólera. Revela que su forma más peligrosa surge gracias a una combinación específica de genes y variantes que le dan ventaja en el intestino humano. Esta investigación podría abrir nuevas vías para predecir y prevenir futuras epidemias de cólera.
El trabajo, publicado en la prestigiosa revista científica PNAS, se centra en Vibrio cholerae, la bacteria causante del cólera. Revela que su forma más peligrosa surge gracias a una combinación específica de genes y variantes que le dan ventaja en el intestino humano. Esta investigación podría abrir nuevas vías para predecir y prevenir futuras epidemias de cólera.
El estudio es fruto de una colaboración entre el investigador de la UMH Mario López Pérez y el profesor Salvador Almagro-Moreno del Hospital St. Jude. Asimismo, han participado el profesor José M. Haro Moreno y la estudiante predoctoral Alicia Campos López, pertenecientes al Departamento de Producción Vegetal y Microbiología de la UMH.
Mediante un análisis exhaustivo de más de 1.840 genomas de Vibrio cholerae, el equipo ha identificado que la especie se divide en once grupos distintos. El grupo pandémico pertenece al más numeroso y está localizado dentro de un linaje compartido con cepas ambientales. Los hallazgos sugieren que las cepas patógenas que causan las epidemias mundiales de cólera emergen sólo cuando se combinan determinadas variaciones alélicas junto con la adquisición de grupos de genes modulares únicos que confieren una ventaja competitiva durante la colonización intestinal. Estas características actúan como un ‘cuello de botella’ crítico que impide que la mayoría de las cepas ambientales se conviertan en patógenos humanos.
“El resultado es que solamente un pequeño grupo de cepas de la bacteria Vibrio cholerae es capaz de causar la enfermedad del cólera en humanos, a pesar de la amplia diversidad de esta especie en la naturaleza”, explica el investigador de la UMH Mario López, primer firmante del estudio, “por lo que nos preguntamos por qué sólo este pequeño grupo ha logrado causar pandemias”.
La investigación revela que la emergencia de las cepas pandémicas del cólera está limitada por ‘cuellos de botella’ genéticos específicos. Se deben dar, a la vez: un fondo genético con adaptaciones previas a la virulencia, la adquisición de genes clave como CTXΦ o VPI-1, su organización en módulos específicos y, finalmente, ciertas variantes alélicas únicas. “Sólo cuando todos estos elementos coinciden, una cepa puede convertirse en un clon patógeno con capacidad pandémica”, explica el profesor.
Estas características no están presentes en otras cepas ambientales de Vibrio cholerae y parecen haber otorgado a las cepas pandémicas una ventaja competitiva clave: una mayor capacidad para colonizar el intestino humano.
“Curiosamente, las características genéticas que permiten a la bacteria del cólera infectar a los humanos no les sirven de nada para prosperar en su medio natural”, explica López. El hábitat natural de Vibrio cholerae son los ambientes acuáticos, donde puede encontrarse por libre o en colonias de cianobacterias, en moluscos o crustáceos.
Como enfermedad, el cólera es endémico en las regiones del mundo con infraestructuras de agua, saneamiento e higiene deficientes. También, se pueden dar brotes tras desastres naturales que afecten a dichas infraestructuras. La infección se caracteriza por episodios severos, repentinos y de gran volumen de diarrea acuosa, que provocan una deshidratación rápida y pueden llevar a la muerte si no se trata de inmediato.
“Nuestro modelo de análisis se podría aplicar a otras bacterias ambientales para entender cómo emergen clones patógenos a partir de poblaciones que, en principio, no lo son”, destaca el experto de la UMH. Además, el estudio abre la puerta a vigilar con más precisión qué cepas tienen potencial pandémico, lo que podría resultar útil para la prevención de futuras crisis sanitarias.
La investigación ha contado con el apoyo de la National Science Foundation (NSF), a través del programa CAREER (#2045671), y de la Burroughs Wellcome Fund, mediante el programa Investigator in the Pathogenesis of Infectious Disease (#1021977). También ha sido financiada por el proyecto español “MICRO3GEN” PID2023-150293NB-I00, cofinanciado con fondos FEDER y gestionado por el Ministerio de Economía, Industria y Competitividad.
Acceso al artículo:
López-Pérez, M., Balasubramanian, D., Campos-Lopez, A., Crist, C., Grant, T. A., Haro-Moreno, J. M., … & Almagro-Moreno, S. (2025). Allelic variations and gene cluster modularity act as nonlinear bottlenecks for cholera emergence. Proceedings of the National Academy of Sciences, 122(22), e2417915122.
Pie de foto: La historia evolutiva del grupo pandémico del cólera en Vibrio cholerae indica que su aparición no ha sido un proceso lineal, sino que ha atravesado varios cuellos de botella clave que explican su rareza. De un grupo de bacterias con determinadas adaptaciones previas a la virulencia (A) surgen otros en los que aparecen clústeres de ciertos genes como CTXΦ o VPI-1 (B), así como módulos específicos dentro de esos clústeres (C) y variaciones alélicas únicas en esos genes (D). La combinación adecuada de todos estos elementos en este mosaico genético permite la aparición de clones patógenos a partir de poblaciones ambientales de V. cholerae (E).
Fuente imagen: López-Pérez et al. (2025), PNAS, 122(22), e2417915122.